 |
|
| Appunti superiori |
|
 |
|
| Appunti superiori |
|
| Visite: 1770 | Gradito: |
Leggi anche appunti:James Hutton (1726-1797)James Hutton (1726-1797) Studiò chimica e medicina, dotato di una straordinaria Fam. Amaryllidaceae (amarillidacee)Fam. Amaryllidaceae (amarillidacee) Comprende circa 100 specie diffuse soprattutto La deriva dei continentiLA DERIVA DEI CONTINENTI La deriva dei continenti è lo spostamento relativo |
 |
 |
TECNICHE DI ANALISI DELLE POPOLAZIONI MICROBICHE
Tutti gli organismi della biosfera dipendono dall attività microbica; in particolare i microrganismi tellurici guidano il ciclo dei nutrienti attraverso la decomposizione, la mineralizzazione, l'immagazzinamento ed il rilascio degli stessi nell'ambiente. Anche nel processo di compostaggio la comunità microbica presente nella sostanza organica solida ha un ruolo chiave. Al fine di migliorare la gestione del processo di compostaggio, è necessario studiare più dettagliatamente le comunità microbiche responsabili dei cambiamenti della materia organica attraverso l analisi della diversità strutturale e funzionale delle stesse e del modo in cui possono rispondere alle variazioni ambientali. Tutte le indagini sui microrganismi presenti nel compost, e più in generale nel suolo, sono ostacolate
dal quantitativo numerico (circa UFC, Unità Formanti Colonia) e dalla
diversità (superiore a 1 taxa distinti) dei microrganismi presenti in un grammo di terreno (Curtis e Sloan, 2005; Gans et al., 2005). Risulta, quindi, determinante l'uso di strumenti che permettano di monitorare e caratterizzare l'ampia diversità microbica di queste matrici complesse.
I metodi per valutare la diversità dei microrganismi del suolo o del compost, sia a livello di specie che di comunità, sono numerosi e solitamente vengono suddivisi in due grandi sottoinsiemi: metodi tradizionali coltura dipendenti e metodi molecolari. Solo in tempi recenti a queste metodologie si sono aggiunte tecniche microscopiche, che consentono lo studio in situ dei microrganismi. Fra queste, ricordiamo la FISH (Fluorescence In Situ Hybridization), che si basa sul rilevamento, mediante l'utilizzo di sonde fluorescenti, di cellule microbiche che presentano specifiche sequenze geniche bersaglio.
1.2.1. Metodi tradizionali coltura dipendenti
I metodi tradizionali rappresentano tutte quelle tecniche che fino agli anni
90 erano le sole utilizzate per studiare la diversità delle popolazioni microbiche presenti nel suolo. Si basano sulla determinazione e sull analisi comparativa di caratteri fenotipici, e quindi, sono confinati a quei microrganismi che possono essere coltivati e isolati in coltura pura.
Tra essi vi sono i metodi basati sull isolamento di microrganismi in terreni di coltura selettivi, che consentono di individuare le Unità Formanti Colonia; allo scopo di massimizzare il recupero dei diversi gruppi microbici, è stata progettata un ampia varietà di mezzi.
Fanno parte di questa tipologia anche i metodi che prevedono la coltivazione su suoli arricchiti, al fine di valutare specifiche attività metaboliche. Pare però che non ci sia garanzia sulla pertinenza tra l attività misurata in laboratorio e quella che avviene nel terreno (Hirsch et al., 2010).
Tra le tecniche tradizionali, sono presenti anche metodologie che analizzano esclusivamente le caratteristiche metaboliche dei microrganismi, come il sistema Biolog . Questo si basa sull'utilizzo di una piastra da 6 pozzetti, che permette di saggiare l'attività catabolica di un ceppo o di una popolazione microbica attraverso la determinazione di profili sperimentali di utilizzo di singole fonti carboniche (Community Level Physiological Profile, CLPP). Nel 1991 Garland e Mills hanno adattato questo sistema allo studio dei profili metabolici di ceppi o comunità complesse, sviluppando delle piastre denominate Eco plate , che contengono fonti di carbonio differenti e rintracciabili in natura. Questo metodo è stato usato con successo per valutare la diversità metabolica potenziale delle comunità microbiche in siti contaminati (Derry et al., 1998; Konopka et al.,
1998), nella rizosfera di alcune piante (Ellis et al., 1995; Garland, 1996; Grayston et al., 1998), nei suoli artici (Derry et al., 1999), nei suoli trattati con erbicidi (El Fantroussi et al., 1999) e nell'inoculazione di microrganismi nel suolo (Bej et al., 1991).
Attraverso le tecniche coltura dipendenti, la conoscenza e la comprensione delle dinamiche ecologiche che si instaurano tra le comunità microbiche rimangono limitate. Il limite più grande è rappresentato dal fatto che tali metodologie consentono di rilevare soltanto i microrganismi coltivabili, e quindi, permettono di studiare solamente quei microrganismi che sono in grado di crescere alle condizioni di incubazione e in presenza di determinati substrati. Questi metodi sono incapaci di rilevare i microrganismi vitali ma non coltivabili (VBNC); pertanto, i risultati ottenuti con tale metodo potrebbero non riflettere la reale diversità funzionale della comunità microbica edafica. È noto, infatti, che la frazione di microrganismi presenti in natura refrattari alla coltivazione è molto ampia: almeno il 99% dei batteri osservati al microscopio non può essere coltivato con tali tecniche (Skinner et al., 1952) e solo una ridotta frazione di funghi presenti nel terreno può essere coltivata con questi metodi di laboratorio Thorn,
1997; Van Elsas et al., 2000; Garbeva et al., 2004). Le cause sono molteplici: una è sicuramente la mancanza di conoscenza delle condizioni in cui la maggior parte dei microrganismi si sviluppa nel proprio ambiente naturale e di conseguenza l'incapacità di costituire un terreno di coltura adatto alla proliferazione delle colonie batteriche e fungine; un altra ragione può essere identificata nell'interdipendenza di organismi diversi gli uni dagli altri, come, per esempio, i microrganismi che possono crescere solo in simbiosi con altri microrganismi o con le piante. La crescita di alcuni microrganismi, inoltre, può essere inibita da sostanze secrete da altri microrganismi presenti nel campione analizzato, o sopraffatta da microrganismi a crescita più veloce che competono per l'utilizzo del substrato. Le tecniche basate sulla coltivazione sono, infine, sensibili alla densità d'inoculo; ad esempio, alcuni studi effettuati sulle comunità fungine hanno mostrato che è favorito lo sviluppo dei funghi che producono una grande quantità di spore, a discapito delle altre specie presenti nel campione (Garland e Mills, 1991).
1.2.2. Metodi molecolari
Negli ultimi venti anni, per superare i problemi associati ai microrganismi non coltivabili, sono state sviluppate varie tecniche molecolari coltura- indipendenti, che consentono di studiare le comunità microbiche nel loro insieme, senza dover isolare ed identificare le singole specie. Tali metodologie si basano principalmente sull'analisi degli acidi nucleici, ma permettono anche lo studio degli acidi grassi di membrana o di altre molecole organiche. Si parla rispettivamente di metodi genetici e di metodi biochimici.
La maggior parte delle metodologie genetiche si avvalgono della reazione a catena della polimerasi (Polymerase Chain Reaction, PCR), una tecnica che ha favorito lo studio della struttura e della dinamica delle popolazioni microbiche presenti nelle diverse matrici ambientali, in quanto consente di analizzare più campioni contemporaneamente e in tempi brevi.
1.2.2.1. Metodi biochimici
Tra i metodi biochimici indipendenti dalla coltivazione dei microrganismi, quelli maggiormente utilizzati per caratterizzare la composizione della comunità microbica sono l'analisi degli esteri metilici degli acidi grassi (Fatty Acid Methyl Ester, FAME) e l'analisi dei fosfolipidi degli acidi grassi (Phospholipid Fatty- acid Analysis, PLFA). Gli acidi grassi costituiscono una percentuale relativamente costante della biomassa cellulare e sono specifici per gruppi di microrganismi, riescono cioè a distinguere i principali gruppi tassonomici all interno di una comunit ; un cambiamento nel profilo degli acidi grassi rappresenta, perciò, un cambiamento nella popolazione microbica.
I metodi basati sull'analisi FAME possono contribuire all'identificazione del microrganismo se effettuati su una coltura pura, mentre possono fornire indicazioni utili a definire la composizione di una comunità microbica se condotti su un campione ambientale. Questa tecnica prevede l'estrazione diretta degli acidi grassi dal campione, la loro metilazione per aumentare la volatilità e infine la separazione mediante gas cromatografia. I profili dei diversi campioni analizzati possono poi essere confrontati tra loro mediante analisi statistiche opportune, come l analisi multivariata.
Il metodo FAME è stato ed è ampiamente utilizzato in ecologia microbica per studiare la composizione delle comunità microbiche ed i loro cambiamenti, indotti da pratiche agricole sul suolo (Esperschutz et al., 2007; Steinbeiss et al.,
2009) e da contaminazione con agenti chimici (Zhang et al., 2006; Kelly et al.,
2007). La tecnica FAME è stata utilizzata anche per misurare la qualità di vari suoli (Winding et al., 2005; Mele e Crowley, 2007).
I fosfolipidi sono acidi grassi caratterizzati da varie catene aciliche, che costituiscono la membrana cellulare dei batteri e degli eucarioti. Gli stessi possono essere estratti direttamente dal suolo e rappresentano solo la componente microbica vitale, in quanto subiscono una rapida degradazione in seguito a morte cellulare.
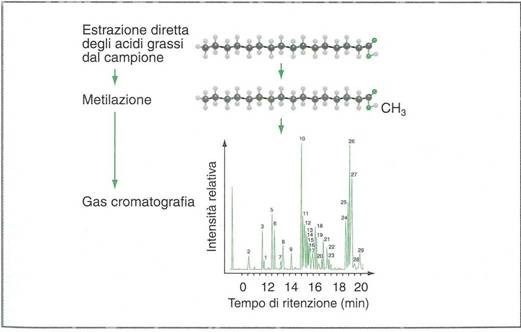
Figura 4: Schema della procedura di analisi FAME Fani et al , 2008)
L'approccio PLFA è stato utilizzato per monitorare la composizione delle comunità microbiche presenti in vari ambienti, tra cui suoli agricoli (Zelles et al.,
1992; Reichardt et al., 1997; Bossio et al., 1998) e torbiere (Sundh et al., 1997). Molti autori si sono avvalsi dell analisi PLFA per caratterizzare i microrganismi presenti nel compost, dimostrando che durante il processo di compostaggio avvengono rapidi cambiamenti delle popolazioni microbiche presenti (Hellmann et al., 1997; Herrmann e Shann, 1997; Carpenter et al., 1998; Klamer e Baath,
1998; Eiland et al., 2001; Sundh e Rönn, 2002).
1.2.2.2. Metodi genetici
I metodi genetici includono tutte le tecniche che studiano le popolazioni microbiche basandosi sugli acidi nucleici, i quali vengono estratti direttamente dai campioni ambientali. Alcune di queste tecniche analizzano il DNA totale, che include anche la componente microbica non più vitale; altre invece studiano l'RNA, che è sintetizzato soltanto dalle cellule in attiva crescita e si degrada rapidamente una volta prodotto, consentendo di identificare solo i membri attivi della comunità microbica in esame.
Nei procarioti, l'RNA messaggero (mRNA) ha spesso una vita molto breve e permette di individuare quali geni siano attivi al momento dell'estrazione.
Tuttavia molte tecniche genetiche si basano sull'analisi dell'RNA ribosomiale (rRNA), una molecola essenziale per la sintesi proteica e che si conserva in tutti i domini filogenetici. Dei vari geni che codificano per l'RNA ribosomiale, l'rDNA 1 S e 23S nei procarioti e l'rDNA 18S e 26S negli eucarioti rappresentano le molecole di elezione per lo studio della diversità microbica a livello di specie e o di genere. Il motivo di base risiede nel fatto che la filogenesi ricostruita utilizzando questi particolari marcatori genetici riflette la storia evolutiva dei microrganismi; inoltre, questi geni mutano molto lentamente durante l'evoluzione e solo in rarissimi casi sono soggetti ad eventi di trasferimento genetico orizzontale. Anche la loro conformazione favorisce l'analisi della comunità microbica mediante amplificazione tramite PCR: sono costituiti da regioni altamente conservate, identiche in tutti gli organismi viventi, e da regioni variabili, che differiscono tra i vari organismi. Ad esempio, la regione V V dell'rDNA 16S Nei batteri e la regione D D2 dell'rDNA 26S negli eucarioti sono zone iper-variabili che danno buone indicazioni circa l affiliazione filogenetica.
I ricercatori hanno pertanto depositato nelle banche dati un numero enorme di sequenze di rRNA batterico e successivamente anche eucariotico, in modo da rendere questi geni marcatori ideali nel confronto tra un ceppo incognito e la banca dati di tutti i microrganismi noti, aprire la strada all'identificazione di microrganismi non coltivabili, e ottenere informazioni tassonomiche sulla comunità microbica totale.
Contenuto in guanina e citosina G + C)
Zhang e Xu (2008) hanno studiato la diversità delle comunità microbiche dei suoli misurando il contenuto in guanina e citosina (G + C) del DNA totale estratto. Questo metodo si basa sul fatto che il DNA dei microrganismi differisce nel contenuto in G + C e che gruppi tassonomicamente vicini differiscono solo per il 3 5% (Tiedje et al., 199 ).
Si tratta di una tecnica quantitativa, in grado cioè di rilevare anche le specie meno abbondanti all interno di una comunit ; essa, tuttavia, richiede grandi quantità di DNA, una tecnica di estrazione del DNA efficiente, oltre ad offrire un livello di risoluzione grossolano.
DNA microarray
La tecnica DNA microarray" sfrutta l'ibridazione DNA-DNA per rilevare ed identificare le specie batteriche o per valutare la diversità microbica. Consiste nell'utilizzo di chip, cui sono adesi covalentemente migliaia di acidi nucleici che funzionano come sonde per geni o sequenze specifiche; il DNA estratto dal ceppo batterico o direttamente dal campione ambientale è marcato con dei fluorocromi ed è fatto ibridare con le sonde fissate sul microarray. Con questo metodo è possibile, quindi, analizzare simultaneamente l espressione di migliaia di sequenze di DNA e ad oggi rappresenta uno degli strumenti di studio molecolare più efficaci. Inoltre, se al posto del DNA viene estratto l'RNA, questo fornisce indicazioni esclusivamente sulla componente attiva della comunità microbica.
In un contesto microbiologico ambientale vengono utilizzati microarrays in cui possono essere presenti collezioni di geni funzionali, collezioni di geni marcatori tassonomici ovvero interi genomi di ceppi di riferimento.
Nel primo caso si parla di GeoChip" (sviluppato da Jizhong Zhou all'Università dell'Oklahoma, USA), le cui sonde permettono di identificare i geni che codificano per enzimi con ruoli chiave nei vari processi ecologici. Sono stati sviluppati vari microarrays di questo tipo, al fine di individuare i microrganismi coinvolti nei cicli biogeochimici (Cho e Tiedje, 2001; Wu et al., 2001; Taroncher- Oldenburg et al., 2003; Steward et al., 2 04; Tiquia et al., 2004); fra le principali attività metaboliche studiate ricordiamo la nitrificazione, la denitrificazione, la fissazione dell'azoto, l'ossidazione del metano, la riduzione dei solfati e la degradazione degli inquinanti.
Nel secondo caso si parla di PhyloChip" (sviluppato da Gary Andersen et al. al Lawrence Berkeley National Lab, USA), poiché i microarrays si basano su sequenze di geni marcatori filogenetici, come i geni che codificano per l'RNA ribosomiale. Tali chip consentono sia di individuare uno specifico microrganismo, sia di studiare la diversità e la struttura delle comunità microbiche presenti nell'ambiente. Negli ultimi anni, sono stati sviluppati microarrays per lo studio dei batteri zolfo riduttori (Small et al., 2001) e dei batteri appartenenti principalmente agli Alphaproteobacteria Sanguin et al., 2005); per il rilevamento delle specie appartenenti a Candida e Aspergillus Leinberger et al., 2005), per quelle appartenenti a Fusarium (Nicolaisen et al., 2005) e per le specie che solitamente sono presenti nel compost Franke Whittle et al , 2005).
Nel terzo caso infine i microarrays sono utilizzati per valutare le differenze nel contenuto genomico tra un ceppo di riferimento e un ceppo naturale isolato dall'ambiente, principalmente allo scopo di analizzare l'espressione genica e comprenderne la regolazione (De Risi et al., 1997; Wodicka et al., 1997; Ye et al.,
2000).
Il limite più grande di questa tecnologia è la necessità di disporre di sequenze di DNA note. I microarrays pertanto non consentono di identificare gruppi non ancora conosciuti e inoltre non sono utilizzabili per lo studio delle comunità fungine, in quanto esistono poche informazioni sulla diversità tassonomica e funzionale delle sequenze geniche dei funghi presenti nel suolo (Hirsch et al., 2010).
Metodi basati sulla PCR
La maggior parte delle tecniche molecolari è basata sull'utilizzo della reazione a catena della polimerasi (PCR), che consente di amplificare frammenti di acidi nucleici compresi tra regioni a sequenza nota. È una tecnica sensibile, che permette di analizzare più campioni contemporaneamente, favorendo l analisi di campioni prelevati da matrici complesse.
Mediante l'amplificazione dei geni codificanti l'rRNA 16S o l'rRNA S e la loro successiva analisi, è possibile ottenere informazioni tassonomiche sui microrganismi coltivabili e non coltivabili; infatti, l utilizzo di primers universali o gruppo specifici, che si legano in regioni del genoma più o meno conservate, permette di rilevare la presenza di particolari organismi o taxa all interno del campione analizzato. Se il prodotto di amplificazione è stato ottenuto da un isolato in coltura pura, l'analisi ARDRA (Amplified Ribosomal DNA Restriction Analysis) o l'analisi della sequenza permetteranno di ottenere informazioni di carattere tassonomico mediante il confronto con ceppi di riferimento o con le sequenze depositate in banca dati. Se invece il prodotto di amplificazione è stato ottenuto dal DNA estratto da una comunità microbica, esso sarà costituito da una miscela eterogenea di molecole di rDNA 16S 18S di diversa provenienza tassonomica. La loro separazione può essere effettuata clonando direttamente l'intero prodotto di amplificazione in appositi vettori plasmidici (librerie di cloni), oppure mediante metodi di fingerprinting" molecolare, tra cui T RFLP, DGGE TGGE, SSCP, RISA ARISA.
Le tecniche molecolari basate sulla PCR hanno quindi favorito lo studio delle comunità microbiche in matrici complesse, pur non essendo esenti da limitazioni. Si elencano di seguito le principali problematiche riscontrate con l'uso di questa tecnica:
la difficoltà di estrazione del DNA da ceppi batterici dotati di involucri esterni particolarmente robusti;
la presenza di più copie di operoni ribosomiali all'interno della cellula, che genera una sovrabbondanza di molecole di rDNA 16S anche in presenza di poche cellule di quella specie nella comunità (Farelly et al.,
1995);
una sottostima della reale diversità microbica, in quanto è favorita l'amplificazione delle specie numericamente dominanti;
la possibilità di formazione di sequenze di rDNA S S "chimeriche", che suggeriscono la presenza di un organismo che nella realtà non esiste Liesack et al., 1991; Kopczynski et al., 1994);
la possibilità di ottenere falsi negativi, generati da impurità presenti nel materiale genetico estratto.
Quest ultimo problema, è particolarmente evidente nell amplificazione del DNA estratto da campioni di suolo. Infatti, per la complessità della matrice suolo, durante l estrazione del DNA, si può avere la coestrazione di sostanze organiche (acidi umici, acidi fulvici, detriti cellulari, proteine) ed inorganiche (metalli pesanti, minerali argillosi) che possono compromettere le analisi post PCR. Ad esempio, gli acidi umici sono in grado di inibire la Taq Dna polimerasi nella reazione di amplificazione chelando gli ioni Mg2+ (Tsai e Olson, 1991; Tebbe e Vahjen, 1993; Miller et al., 1999). Alcuni autori hanno risolto il problema effettuando una diluizione del DNA estratto, oppure aggiungendo degli agenti in grado di sequestrare gli inibitori come la BSA direttamente nella miscela di reazione Tebbe e Vahjen, 1993; Yeates et al., 1997). In molti casi, comunque, anche una diluizione molto spinta ( 0 volte) non riesce ad eliminare l'effetto PCR inibitore degli acidi umici coestratti (Erb e Wagnerdöbler, 1993). Da qui la necessità di purificare il materiale genetico estratto anche quando ciò comporta perdite significative di DNA. Le tecniche più ampiamente applicate per la purificazione del DNA estratto dal suolo si basano su ultracentrifugazione in gradiente di densità di cesio cloruro, cromatografia a scambio ionico, elettroforesi in gel d'agarosio, o ancora su dialisi (Roose-Amsaleg et al., 2001). Alcuni di questi metodi possono richiedere delle ottimizzazioni, specie se applicati a campioni particolarmente ricchi di sostanza organica come il compost Tsai e Olson, 1991; Zhou et al., 1996; Harry et al., 1999).
Recentemente, sono stati sviluppati metodi di estrazione che fanno uso di kit commerciali specifici per l'estrazione diretta di DNA dal suolo. I più diffusi sono l'UltraCleanSoilT DNA kit ed il PowerSoilT DNA Isolation Kit Sample della MOBIO, ed il FastDNA® SPIN Kit for Soils della BIO101 Inc. Il materiale ottenuto con tali metodi è solitamente utilizzabile per la PCR senza la necessità di ulteriori fasi di purificazione.
Librerie di cloni
La separazione degli amplificati provenienti dai diversi microrganismi avviene mediante il clonaggio degli stessi in vettori plasmidici inseriti in cellule competenti di Escherichia coli. Il prodotto ottenuto è una libreria di molecole di rDNA 16S o 18S che vengono sequenziate singolarmente; le sequenze ottenute sono così utilizzate per recuperare dalle banche dati le sequenze ad esse più simili, con le quali vengono allineate.
Questa strategia permette di ottenere una stima della diversità tassonomica esistente nel campione analizzato. Blanc et al. (1999), ad esempio, hanno studiato una libreria di cloni di geni di rRNA 16S, al fine di caratterizzare la comunità batterica presente nella fase termofila del processo di compostaggio. Tuttavia, per descrivere accuratamente la diversità microbica in un campione complesso, sono necessarie librerie di cloni abbastanza grandi, ma sono ancora pochi gli studi che hanno analizzato in modo soddisfacente la questione della rappresentatività (Lauber et al., 2008).
T-RFLP (Terminal Restriction Fragment Length Polymorphism)
La tecnica T-RFLP è un'analisi del polimorfismo della lunghezza dei frammenti terminali di restrizione dei geni d'interesse (di solito l'rDNA 16S), basata sulla digestione con endonucleasi di restrizione degli amplificati marcati all'estremità con una molecola fluorescente, separati successivamente mediante elettroforesi su gel o su capillare. Il risultato di tale analisi è un elettroferogramma costituito da una serie di picchi che differiscono per mobilità (taglia molecolare), altezza e area, e che rappresentano le diverse forme microbiche presenti nella comunità in esame. Ogni specie produrrà un solo frammento "visibile" e quindi il numero dei frammenti nel profilo sarà direttamente proporzionale al numero di diverse specie microbiche presenti nel campione (Fani et al., 2008). I profili di picchi di campioni ambientali diversi possono essere comparati tra loro in modo da evidenziare similarità e differenze tra le comunità microbiche in esame.
L'altezza di ogni picco dovrebbe fornire una misura della proporzione relativa di ogni componente della popolazione. Tuttavia, a causa dell'annealing preferenziale del primer su alcuni stampi di DNA rispetto ad altri (Suzuki e Giovannoni, 1996), il valore assoluto di questa misura deve essere trattato con cautela. Inoltre, microrganismi differenti possono avere, per un determinato enzima, un frammento di restrizione terminale (TRF) della stessa dimensione e quindi non essere distinguibili in base ad un singolo profilo di restrizione (Tiedje et al., 1999). Questo è particolarmente vero per i TRF generati dall'estremità 3' terminale del gene dell'rDNA 16S; una discriminazione maggiore è fornita dalla regione 5', poiché le regioni V1, V2 e V3 presenti su questa estremità sono più eterogenee (Suzuki et al., 1998). Per caratterizzare un microrganismo, solitamente si utilizzano i profili T-RFLP ottenuti con quattro diversi enzimi di restrizione.
Nonostante la tecnica T-RFLP soffra delle stesse limitazioni comuni a tutte le analisi basate sulla PCR, risulta una delle tecniche più utilizzate al momento in microbiologia ambientale per l'analisi filogenetica delle comunità microbiche, anche in combinazione con altre metodologie. Infatti, è facile sia nella messa a punto che nell'uso, può essere in parte automatizzata e permette di confrontare un numero elevato di campioni, analizzati anche in momenti diversi.
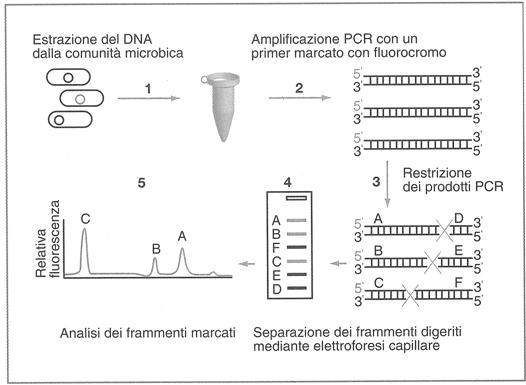
Figura 5: Schema della procedura T-RFLP applicata all'analisi molecolare di una comunità microbica (Fani et al., 2008)
Esiste una vasta letteratura sull'utilizzo del T-RFLP come strumento di studio delle comunità microbiche presenti nell'ambiente. In particolare, l'analisi T-RFLP rappresenta uno strumento molto utile per valutare i cambiamenti nella struttura della comunità microbica presente in bioreattori sviluppati per la degradazione di policlorobifenili (Fedi et al., 2005) e in reattori utilizzati per il biorisanamento di reflui agro-industriali ricchi in composti fenolici (Bertin et al.,
2006). Sono state studiate comunità microbiche complesse mediante l'analisi dei profili T-RFLP da molti autori, tra i quali citiamo Hartmann e Widmer (2006) e Liu et al. (1997), che hanno dimostrato le elevate potenzialità di questa tecnica nella valutazione della diversità delle comunità batteriche complesse e nel confronto della struttura e della diversità delle comunità presenti in ecosistemi diversi.
Lo studio delle comunità batteriche e fungine mediante T-RFLP in suoli fertilizzati con compost ha rivelato la presenza di popolazioni differenti rispetto a quelle presenti nel suolo non fertilizzato (Pérez-Piqueres et al., 2006). Tiquia (2005) ha studiato la diversità delle comunità batteriche e fungine del compost a differenti stadi del processo di compostaggio. Sono state rilevate variazioni delle comunità durante il processo ed è stato osservato che le comunità batteriche hanno una diversità maggiore rispetto a quelle fungine. Lo stesso studio è stato svolto da Székeley et al. (2008), che hanno utilizzato sia la tecnica T-RFLP sia la DGGE, ottenendo con entrambi i metodi profili quasi identici.
RISA (Ribosomal Intergenic Spacer Analysis) / ARISA (Automated Ribosomal
Intergenic Spacer Analysis)
La RISA è un'altra tecnica di DNA fingerprinting, simile in linea di principio al T-RFLP e basata sul polimorfismo di lunghezza della regione spaziatrice compresa tra i geni codificanti per l'rRNA 16S e 23S (ITS, Internal Transcribed Spacer). I primers sono disegnati su regioni relativamente conservate così da garantire l'amplificazione da un ampio spettro tassonomico; la regione ITS, che viene amplificata mediante PCR e separata su gel di poliacrilammide in condizioni denaturanti, ha un'elevata eterogeneità di lunghezza e di sequenza e permette di differenziare tra ceppi batterici e specie strettamente correlate (Fisher e Triplett, 1999). I polimorfismi sono poi rilevati utilizzando la colorazione argentica.
La RISA è stata applicata con successo negli studi sulla diversità della comunità microbica nel terreno (Borneman e Triplett, 1997), nella rizosfera delle piante (Borneman e Triplett, 1997) e nei suoli contaminati (Ranjard et al., 2000).
Come evoluzione logica della RISA nasce l'ARISA, una tecnica sviluppata da Fisher e Triplett (1999), basata sull'utilizzo di un primer marcato con fluorocromo. I prodotti di PCR sono analizzati mediante un sistema di elettroforesi capillare automatizzato che produce un elettroferogramma, i cui picchi corrispondono ai frammenti di DNA marcati terminalmente. La sensibilità della tecnica è maggiore rispetto alla RISA, si riscontra un'elevata semplicità di lettura dei dati ottenuti e la riproducibilità è garantita dall'automazione dello strumento. Queste caratteristiche rendono l'ARISA una metodologia attendibile per l'analisi ed il confronto di un gran numero di campioni, anche se è ancora soggetta alle tradizionali restrizioni della PCR.
L'ARISA è stata utilizzata per analizzare la struttura genetica di diverse comunità batteriche e fungine presenti in differenti tipi di suolo (Ranjard et al., 2001) e per valutare i cambiamenti delle comunità batteriche (Schloss et al., 2003) e fungine (Hansgate et al., 2005) nelle fasi iniziali del processo di compostaggio.
Estrazione del DNA
Confronto tra i profili dei diversi campioni
Amplificazion e mediante PCR dell'ITS con primer
marcati Ampliconi di lunghezza
variabile
Elettroforesi su gel o capillare dei frammenti amplificati
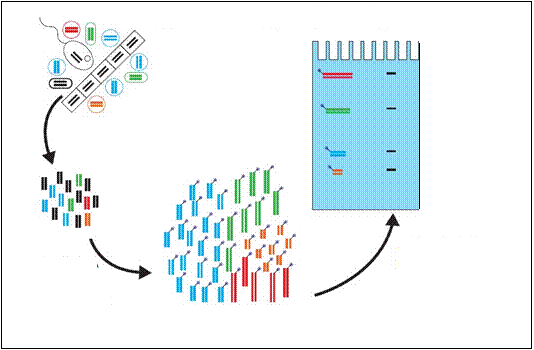 Figura 6: Schema della procedura ARISA (Byron, 2005)
Figura 6: Schema della procedura ARISA (Byron, 2005)
SSCP (Single Strand Conformation Polymorphism)
La tecnica SSCP individua i polimorfismi di conformazione delle singole eliche sfruttando la tendenza del DNA a singolo filamento ad assumere conformazioni specifiche, strettamente dipendenti dalla sequenza nucleotidica, in relazione ai legami intramolecolari che si instaurano tra le basi complementari. La mobilità elettroforetica di una molecola di DNA a singolo filamento cambia in funzione delle sue dimensioni, della sua sequenza nucleotidica, della temperatura di analisi e della forza ionica. Perciò, in condizioni non denaturanti e a parità di temperatura, la mobilità elettroforetica di un singolo filamento di DNA varia in base alla sua conformazione intramolecolare (Lee et al., 1996). I singoli filamenti di DNA ottenuti mediante amplificazione in PCR, denaturati e sottoposti a rapido raffreddamento, vengono fatti migrare su un gel di poliacrilammide in condizioni non denaturanti. I frammenti di DNA che presentano alterazioni anche di una singola base vengono evidenziati per un cambiamento nella loro mobilità elettroforetica e si identificano mediante colorazione argentica.
Il metodo è economico e versatile, anche se si rileva che la sensibilità è limitata dalle dimensioni del frammento e tende a diminuire con l'aumentare della lunghezza dei frammenti di DNA. Inoltre, alcuni DNA a singolo filamento possono formare più di una conformazione stabile, cosicché una sequenza può essere rappresentata da più di una banda sul gel (Tiedje et al., 1999).
La tecnica SSCP è stata utilizzata in ecologia microbica per studiare la diversità sia delle comunità batteriche che fungine in varie matrici ambientali, tra cui la rizosfera (Schwieger e Tebbe, 1998; Schmalenberger et al., 2001). Peters et al. (2000) hanno utilizzato con successo la tecnica SSCP per caratterizzare la diversità e la successione delle comunità microbiche durante il processo di compostaggio di residui agricoli.
DGGE (Denaturing Gradient Gel Electrophoresis) / TGGE (Thermal Gradient
Gel Electrophoresis)
La DGGE e la TGGE sono due metodologie molto simili, sviluppate per la ricerca di mutazioni puntiformi in genetica medica; successivamente il loro utilizzo è stato allargato allo studio della diversità genetica microbica (Muyzer et al., 1993)
Per mezzo di una corsa elettroforetica su un gel di poliacrilammide contenente un gradiente lineare di agenti denaturanti di tipo chimico, costituito da una miscela di urea e formammide nel caso della DGGE oppure da un gradiente denaturante di tipo termico nella TGGE, è possibile separare frammenti di DNA amplificati mediante PCR, aventi cioè la stessa lunghezza, ma differente sequenza. Teoricamente, queste tecniche sono capaci di separare sequenze nucleotidiche che differiscono per una sola coppia di basi. Nübel et al. (1996) hanno separato frammenti di DNA provenienti da diversi operoni rrN, alcuni dei quali differivano per una sola base, mentre Kowalchuk et al. (1997) hanno dimostrato che le doppie bande rilevate nei profili DGGE da loro studiati, erano il risultato della cosiddetta wobble base, l'oscillazione di una citosina o di una timina nel primer reverse.
Le due tecniche si basano sul principio molecolare per il quale gli ampliconi, durante la corsa, incontrano un gradiente denaturante sempre maggiore, che genera la parziale separazione dei filamenti costituenti la doppia elica. Infatti, la denaturazione delle parti della molecola aventi una temperatura di melting minore causa una riduzione della velocità di migrazione dell'intero frammento, fino ad una quasi completa stabilizzazione. Questo punto di parziale denaturazione è, in teoria, diverso da molecola a molecola in relazione alla specifica sequenza di basi (Muyzer et al., 1993; Heuer e Smalla, 1997).
Per una migliore separazione fisica spaziale delle molecole con sequenza differente, si utilizza, durante l'amplificazione, un primer forward contenente in
5' una breve sequenza di 40-50 pb a doppia elica, ricca in basi G e C (Myers et al., 1987). Questa coda-GC ha una resistenza maggiore alle condizioni denaturanti e consente di tenere uniti i frammenti di DNA che nella successiva corsa elettroforetica si denatureranno completamente.
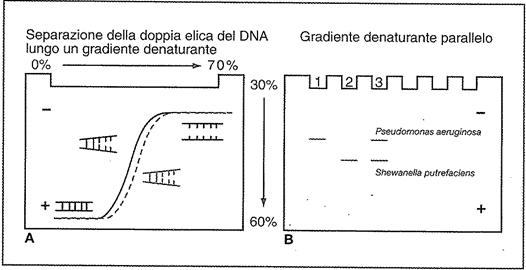
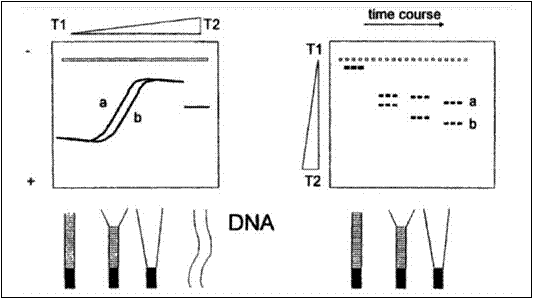
Figura 7: Schema del processo di DGGE: in A il gradiente denaturante separa la doppia elica del DNA e ne cambia la velocità di migrazione attraverso le maglie del gel. In B nei pozzetti 1 e 2 sono mostrate le bande del 16S rDNA che corrispondono a due specie batteriche diverse. A causa del loro differente contenuto in G + C la velocità di migrazione sarà diversa, permettendo una loro separazione in un campione che le contenga entrambe (pozzetto 3) (Fani et al., 2008)
Le bande che si ottengono nei profili DGGE e TGGE possono essere rilevate mediante colorazione con bromuro d'etidio oppure utilizzando il SYBR Green I. Sono entrambi agenti che si intercalano nel DNA a doppio filamento, anche se il SYBR Green I ha una sensibilità circa 25 volte maggiore rispetto a quella del bromuro d'etidio. Un metodo di rilevamento ancora più sensibile è la colorazione argentica (Felske et al., 1996). Tuttavia questo colorante si lega anche al DNA a singolo filamento, impedendo un'eventuale analisi d'ibridazione successiva (Heuer e Smalla, 1997).
La TGGE può essere condotta su gel con gradiente di temperatura perpendicolare o parallelo al campo elettrico. Allo stesso modo, la DGGE può essere condotta su gel con gradiente denaturante perpendicolare o parallelo al campo elettrico. Nel primo caso, generalmente l'ambito del gradiente è ampio (0-
 100% oppure 20-100%); nel secondo caso, l'ampiezza del gradiente è più ristretta e consente una migliore separazione degli ampliconi (Myers et al., 1987). Quest'ultimo tipo
di
gel è comunemente più utilizzato, perché permette il
caricamento contemporaneo di più campioni.
100% oppure 20-100%); nel secondo caso, l'ampiezza del gradiente è più ristretta e consente una migliore separazione degli ampliconi (Myers et al., 1987). Quest'ultimo tipo
di
gel è comunemente più utilizzato, perché permette il
caricamento contemporaneo di più campioni.
Figura 8: Schema del profilo TGGE su gel perpendicolare (a sinistra) e parallelo (a destra) di due diversi ceppi. Il frammento di DNA colorato in nero rappresenta la coda-GC (Heuer e Smalla,
1997).
Sia la DGGE che la TGGE hanno il vantaggio di essere affidabili, riproducibili, rapide e non molto costose. Inoltre, la tecnica DGGE permette di analizzare contemporaneamente più campioni prelevati ad intervalli di tempo diversi, rendendola idonea per gli studi sull'evoluzione delle popolazioni microbiche nell'ambiente.
I limiti di questa metodologia relativi ai passaggi operativi antecedenti la tecnica stessa possono essere il campionamento di una matrice non correttamente omogeneizzata o la variabilità di estrazione del DNA, che non garantiscono la rappresentatività della matrice analizzata. Inoltre, essendo basata sulla PCR, è soggetta alle restrizioni di questa tecnica.
Le limitazioni proprie della tecnica DGGE riguardano vari aspetti:
. questa tecnica non permette la separazione di frammenti di lunghezza superiore a 600 pb circa, limitando così la scelta delle regioni da analizzare;
. non sono rilevabili le specie numericamente inferiori, soprattutto nel suolo, dove la diversità è molto alta. Heuer e Smalla (1997) hanno comunque dimostrato che un ceppo rappresentante l'1% della popolazione batterica totale è efficacemente rilevato, come banda distinta, in un profilo DGGE;
. i frammenti di DNA di sequenza differente potrebbero avere la stessa mobilità nel gel di poliacrilammide e quindi, una banda potrebbe non necessariamente rappresentare una sola specie (Gelsomino et al., 1999);
. si possono ottenere bande multiple per una stessa specie batterica, a causa della ridondanza di operoni ribosomiali in una cellula, i quali possono presentare microeterogeneità (Nübel et al., 1996);
. prevede, come del resto la TGGE, una laboriosa manipolazione dei campioni che potrebbe influenzare la comunità microbica (Muyzer,
1999).
Ad ogni modo, la DGGE è ,probabilmente, la metodologia più ampiamente utilizzata tra le tecniche di studio delle comunità microbiche presenti nei campioni ambientali, soprattutto in applicazioni di tipo tassonomico. In linea di principio, questa tecnica, analogamente al T-RFLP, può essere applicata a qualsiasi sequenza di DNA amplificato per PCR da un ceppo batterico o da una comunità microbica. Solitamente però è analizzato il gene marcatore del DNA ribosomiale
16S per i batteri e 18S per i microrganismi eucarioti, che hanno importanza filogenetica. Volendo generare profili rappresentanti i batteri metabolicamente più attivi, è possibile utilizzare l'RNA ribosomiale retrotrascritto, anziché il DNA, perché le cellule metabolicamente più attive hanno un contenuto in RNA maggiore rispetto a quelle meno attive (Heuer e Smalla, 1997).
Ad oggi, vi è un'ampia letteratura sull'impiego di queste metodologie in campo microbiologico; in particolar modo le applicazioni della DGGE sono rivolte all'identificazione e alla tipizzazione di forme microbiche mediante il confronto di profili DGGE di microrganismi ignoti e microrganismi isolati in coltura pura (Nielsen et al., 2000; Theelen et al., 2001; Gurtler et al., 2002; Agnelli et al., 2004), ma anche allo studio della struttura e dell'evoluzione di comunità microbiche presenti in varie matrici ambientali, tra cui il suolo (Heuer e Smalla, 1997; Norris et al., 2000; Smalla et al., 2001; Avrahami et al., 2003; Nicol et al., 2003; Crecchio et al., 2007) e il compost (Kowalchuk et al., 1999; Ishii et al., 2000; Pedro et al., 2001; Cahyani et al., 2003; Marshall et al., 2003; Takaku et al., 2006; Novinscak et al., 2009; Zhang et al., 2010; Federici et al.,
2011). Alfreider et al. (2002) hanno studiato le comunità che si sviluppano nel compost, confrontando la tecnica DGGE e la SSCP; entrambi i metodi sono risultati idonei, seppur producevano profili non identici, probabilmente a causa dei diversi primers per l'amplificazione del 16S rDNA. Wang e Xu (2011) hanno invece utilizzato la tecnologia DGGE per confrontare la successione microbica in cumuli in compostaggio, inoculati e non, con alcuni funghi che potrebbero favorire i processi di umificazione, riducendo i tempi di processo.
Recentemente, la tecnica DGGE è stata utilizzata insieme ad altre metodologie biochimiche per valutare l'impatto di matrici organiche tossiche, come la sansa di olive, sulla struttura e sulla diversità delle comunità microbiche che si sviluppano durante il processo di compostaggio di queste matrici (Vivas et al., 2009; Federici et al., 2011).
 |
| Appunti su: |
|